![]() SEGUI, CONTINUA
SEGUI, CONTINUA
Una nuova tecnica innovativa sviluppata da un team scientifico del Center for Genomic Regulation (CRG) di Barcellona ha scoperto l'esistenza di una moltitudine di "telecomandi" che controllano la funzione delle proteine e che possono essere usati come bersagli per ottenere farmaci più efficaci ed efficiente in varie patologie come demenza, cancro e infezioni infettive.
Questi "telecomandi" sono scientificamente conosciuti come siti allosterici. Si tratta di telecomandi che sono distanti dal sito di azione della proteina, ma hanno la capacità di regolarla o modularla", Júlia Domingo, prima coautrice dello studio, pubblicato questo mercoledì sulla rivista "Nature", spiegato all'ABC. E aggiunge una similitudine: "È come se con quel telecomando si potesse accendere e spegnere la lampadina o regolare l'intensità della luce".
In questo caso si intende bloccare o regolare l'attività di proteine che mantengono la loro alterata funzione in confinamento. Ad esempio, nel caso del cancro, le proteine che hanno acquisito una mutazione hanno la loro funzionalità alterata, lo fanno in modo anomalo e la cellula cresce in modo insolito. In molti casi non esistono farmaci in grado di modulare o bloccare questa attività anormale o, se presenti, non sono specifici e vengono rilasciati anche da altre proteine che funzionano normalmente.
Tradizionalmente, i cacciatori di droghe hanno progettato trattamenti che prendono di mira il sito attivo di una proteina, la cui piccola regione produce reazioni chimiche in cui i bersagli si legano. Lo svantaggio di questi farmaci, noti come farmaci ortosterici, è che i siti attivi di molte proteine sono molto simili e i farmaci hanno legato e inibito molte proteine diverse contemporaneamente, anche quelle che funzionano normalmente e non sono interessanti da toccare, che può causare effetti collaterali.
“Lì è entrato nel concetto di allosteria e nel potenziale che ha per progettare farmaci. La cosa interessante dei siti allosterici è che sono super specifici per ogni proteina. Se questi siti allosterici trovano parte della superficie della proteina dove il farmaco può atterrare, sarà estremamente specifico per quella proteina. Potremo aspirare a farmaci più efficaci”, sottolinea il ricercatore.
“Non solo scopriamo che questi siti terapeutici sono abbondanti, ma ci sono prove che possono essere manipolati in molti modi diversi. Invece di accenderli e spegnerli, possiamo modulare la loro attività come un termostato. Da un punto di vista ingegneristico è come se avessimo colpito l'oro, perché ci dà molto spazio per progettare 'droghe intelligenti' che vanno al male e saltano il bene", spiega André Faure, ricercatore post-dottorato al CRG e primo co-autore dell'articolo.
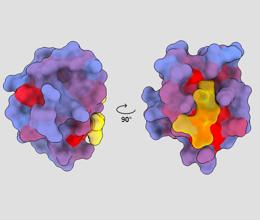 immagine tridimensionale che mostra la proteina umana PSD95-PDZ3 da diversi punti di vista. In giallo è mostrata una molecola che si lega al sito attivo. Il gradiente di colore dal blu al rosso indica possibili siti allosterici: André Faure/ChimeraX
immagine tridimensionale che mostra la proteina umana PSD95-PDZ3 da diversi punti di vista. In giallo è mostrata una molecola che si lega al sito attivo. Il gradiente di colore dal blu al rosso indica possibili siti allosterici: André Faure/ChimeraX
Per questa scoperta, il team ha utilizzato un metodo che consente loro di assumere una forma proteica e sistemica e un incontro globale con tutti i siti. Per fare ciò, hanno scelto due proteine molto abbondanti nel nostro proteoma umano. “Il 50% della superficie delle proteine ha un potenziale allosterico. Il nostro metodo permette di realizzare un atlante di siti allosterici, che renderebbe molto più efficiente il processo di ricerca di farmaci efficaci”, assicura Júlia Domingo.
Gli autori dello studio hanno sviluppato una tecnica chiamata double-depth PCA (ddPCA), che descrivono come un "esperimento di forza bruta". "Rompiamo di proposito le cose in migliaia di modi diversi per formare un quadro completo di come funziona qualcosa", spiega il Professore di ricerca dell'ICREA Ben Lehner, coordinatore del programma di biologia dei sistemi presso il CRG e autore dello studio. “È come se sospetti che una candela sia difettosa, ma invece di limitarsi a controllarla, il meccanico smonta l'intera macchina e controlla tutte le parti una per una. Analizzando diecimila cose contemporaneamente, identifichiamo tutti i pezzi che sono veramente importanti”.
Successivamente, utilizziamo algoritmi di intelligenza artificiale per interpretare i risultati di laboratorio.
Uno dei grandi vantaggi del metodo, oltre a semplificare il processo necessario per trovare siti allosterici, è che è una tecnica economica e accessibile per qualsiasi laboratorio di ricerca nel mondo. “Richiede solo l'accesso ai reagenti di biologia molecolare di base, l'accesso a un sequenziatore di DNA e un computer. Con questi tre componenti, qualsiasi laboratorio in 2-3 mesi, con un piccolo budget, può effettuare questo esperimento sulla proteina di interesse che desidera”, assicura Júlia Domingo. La speranza dei ricercatori è che i nostri scienziati utilizzino la tecnica per mappare in modo rapido e completo i siti allosterici delle proteine umane uno per uno. “Se disponiamo di dati sufficienti, forse un giorno potremo fare un ulteriore passo avanti e prevedere dalla sequenza proteica alla funzione. Usa questi dati per guidarli come terapie migliori per prevedere se un certo cambiamento in una proteina degenererà in una malattia”, ha concluso il ricercatore.