![]() SIGUE
SIGUE
Unha nova técnica innovadora desenvolvida por un equipo científico do Centro de Regulación Xenómica (CRG) de Barcelona descubriu a existencia de multitude de 'mandos a distancia' que controlan a función das proteínas e que poden ser utilizados como dianas para conseguir fármacos máis eficaces. e eficiente en diversas patoloxías como a demencia, o cancro e as infeccións infecciosas.
Estes "controis remotos" coñécense cientificamente como sitios alostéricos. Trátase de mandos a distancia afastados do lugar de acción da proteína, pero que teñen capacidade para regulala ou modulala”, Júlia Domingo, primeira coautora do estudo, que se publica este mércores na revista “Nature”. explicou a ABC. E engade un símil: "É coma se con ese mando a distancia puideses prender e apagar a bombilla ou regular a intensidade da luz".
Neste caso onde se pretende bloquear ou regular a actividade das proteínas que manteñen a súa función alterada en confinamento. Por exemplo, no caso do cancro, as proteínas que adquiriron unha mutación teñen a súa funcionalidade alterada, fano de forma anormal e a célula crece dun xeito inusual. En moitos casos, non existen fármacos que poidan modular ou bloquear esta actividade anormal ou, se os hai, non son específicos e tamén se liberan doutras proteínas que funcionan normalmente.
Tradicionalmente, os cazadores de drogas deseñaron tratamentos que teñen como obxectivo o sitio activo dunha proteína, cuxa pequena rexión produce reaccións químicas onde se unen os obxectivos. O inconveniente destes fármacos, coñecidos como fármacos ortostéricos, é que os sitios activos de moitas proteínas son moi similares e os fármacos uniron e inhibiron moitas proteínas diferentes ao mesmo tempo, incluso aquelas que funcionan normalmente e non son interesantes de tocar, o que pode causar efectos secundarios.
“Alí entrou no concepto de alosteria e no potencial que ten para deseñar fármacos. O interesante dos sitios alostéricos é que son súper específicos para cada proteína. Se estes sitios alostéricos atopan parte da superficie da proteína onde a droga pode aterrar, será moi específica para esa proteína. Poderemos aspirar a medicamentos máis eficaces”, apunta o investigador.
"Non só descubrimos que estes sitios terapéuticos son abundantes, senón que hai evidencias de que poden manipularse de moitas formas diferentes. En lugar de acendelos e apagalos, podemos modular a súa actividade como un termostato. Desde o punto de vista da enxeñaría, é coma se tivemos ouro, porque nos dá moito marxe para deseñar 'smart drugs' que van para o mal e se saltan o bo", explica André Faure, investigador posdoutoral do CRG. e primeiro coautor do artigo.
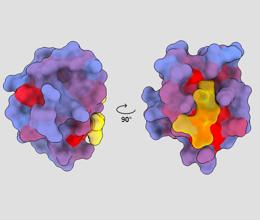 imaxe tridimensional que mostra a proteína humana PSD95-PDZ3 desde diferentes puntos de vista. Unha molécula móstrase uníndose ao sitio activo en amarelo. O gradiente de cor azul a vermello indica posibles sitios alostéricos - André Faure/ChimeraX
imaxe tridimensional que mostra a proteína humana PSD95-PDZ3 desde diferentes puntos de vista. Unha molécula móstrase uníndose ao sitio activo en amarelo. O gradiente de cor azul a vermello indica posibles sitios alostéricos - André Faure/ChimeraX
Para este descubrimento, o equipo utilizou un método que lles permite tomar unha proteína e unha forma sistémica e un encontro global con todos os sitios. Para iso, escolleron dúas proteínas moi abundantes no noso proteoma humano. "O 50% da superficie da proteína ten potencial alostérico. O noso método permite facer un atlas de sitios alostéricos, o que faría moito máis eficiente o proceso de busca de fármacos eficaces”, asegura Júlia Domingo.
Os autores do estudo desenvolveron unha técnica chamada PCA de dobre profundidade (ddPCA), que describen como un "experimento de forza bruta". "Decidimos romper as cousas de miles de formas diferentes para formar unha imaxe completa de como funciona algo", explica o profesor de investigación ICREA Ben Lehner, coordinador do programa de Bioloxía de Sistemas do CRG e autor do estudo. "É como se sospeitas que unha bujía está mal, pero en lugar de comprobar iso, o mecánico desmontará todo o coche e revisará todas as pezas unha por unha. Ao analizar dez mil cousas á vez, identificamos todas as pezas que son realmente importantes”.
A continuación, utilizamos algoritmos de intelixencia artificial para interpretar os resultados do laboratorio.
Unha das grandes vantaxes do método, ademais de simplificar o proceso necesario para atopar xacementos alostéricos, é que é unha técnica asequible e accesible para calquera laboratorio de investigación do mundo. "Só require acceso a reactivos básicos de bioloxía molecular, acceso a un secuenciador de ADN e unha computadora. Con estes tres compoñentes calquera laboratorio en 2-3 meses, cun orzamento reducido, pode realizar este experimento sobre a proteína de interese que queira”, asegura Júlia Domingo. A esperanza dos investigadores é que os nosos científicos usen a técnica para mapear de forma rápida e completa os sitios alostéricos das proteínas humanas un por un. "Se temos datos suficientes, quizais algún día poidamos dar un paso máis e predecir desde a secuencia da proteína ata a función. Utiliza estes datos para orientalos como mellores terapias para predecir se un determinado cambio nunha proteína vai dexenerar nunha enfermidade”, concluíu o investigador.
