![]() SUIVRE
SUIVRE
Une nouvelle technique innovante développée par une équipe scientifique du Centre de régulation génomique (CRG) de Barcelone a découvert l'existence d'une multitude de "télécommandes" qui contrôlent la fonction des protéines et qui peuvent être utilisées comme cibles pour obtenir des médicaments plus efficaces et efficace dans diverses pathologies telles que la démence, le cancer et les infections infectieuses.
Ces «télécommandes» sont scientifiquement connues sous le nom de sites allostériques. Ce sont des télécommandes éloignées du site d'action de la protéine, mais qui ont la capacité de le réguler ou de le moduler", Júlia Domingo, première co-auteur de l'étude, publiée ce mercredi dans la revue "Nature", expliqué à ABC. Et il ajoute une comparaison : "C'est comme si avec cette télécommande on pouvait allumer et éteindre l'ampoule ou régler l'intensité de la lumière."
En l'occurrence où il s'agit de bloquer ou de réguler l'activité de protéines qui maintiennent leur fonction altérée en confinement. Par exemple, dans le cas du cancer, les protéines qui ont acquis une mutation voient leur fonctionnalité altérée, elles le font de façon anormale et la cellule se développe de façon inhabituelle. Dans de nombreux cas, aucun médicament ne peut moduler ou bloquer cette activité anormale ou, s'il y en a, ils ne sont pas spécifiques et sont également libérés par d'autres protéines qui fonctionnent normalement.
Traditionnellement, les chasseurs de drogue ont conçu des traitements qui ciblent le site actif d'une protéine, dont la petite région produit des réactions chimiques où les cibles se lient. L'inconvénient de ces médicaments, connus sous le nom de médicaments orthostériques, est que les sites actifs de nombreuses protéines sont très similaires et que les médicaments ont lié et inhibé de nombreuses protéines différentes en même temps, même celles qui fonctionnent normalement et ne sont pas intéressantes à toucher, ce qui peut provoquer des effets secondaires.
"Là, il est entré dans le concept d'allostérie et le potentiel qu'elle a pour concevoir des médicaments. La chose intéressante à propos des sites allostériques est qu'ils sont super spécifiques pour chaque protéine. Si ces sites allostériques trouvent une partie de la surface de la protéine où le médicament peut atterrir, il sera extrêmement spécifique pour cette protéine. On pourra aspirer à des médicaments plus efficaces », pointe le chercheur.
« Non seulement nous constatons que ces sites thérapeutiques sont abondants, mais il existe des preuves qu'ils peuvent être manipulés de différentes manières. Au lieu de simplement les allumer et les éteindre, nous pouvons moduler leur activité comme un thermostat. D'un point de vue ingénierie, c'est comme si on avait trouvé de l'or, car cela nous laisse une grande marge de manœuvre pour concevoir des "médicaments intelligents" qui vont au mal et sautent le bien", explique André Faure, chercheur postdoctoral au CRG et premier co-auteur de l'article.
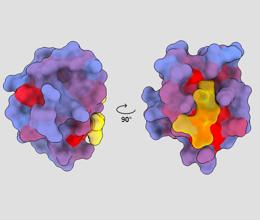 image tridimensionnelle montrant la protéine humaine PSD95-PDZ3 sous différents points de vue. Une molécule est montrée se liant au site actif en jaune. Un dégradé de couleur bleu à rouge indique de possibles sites allostériques – André Faure/ChimeraX
image tridimensionnelle montrant la protéine humaine PSD95-PDZ3 sous différents points de vue. Une molécule est montrée se liant au site actif en jaune. Un dégradé de couleur bleu à rouge indique de possibles sites allostériques – André Faure/ChimeraX
Pour cette découverte, l'équipe a utilisé une méthode qui leur permet de prendre une protéine et une forme systémique et une rencontre globale avec tous les sites. Pour ce faire, ils ont choisi deux protéines très abondantes dans notre protéome humain. « 50 % de la surface protéique a un potentiel allostérique. Notre méthode permet de faire un atlas des sites allostériques, ce qui rendrait beaucoup plus efficace le processus de recherche de médicaments efficaces », assure Júlia Domingo.
Les auteurs de l'étude ont développé une technique appelée PCA à double profondeur (ddPCA), qu'ils décrivent comme une "expérience de force brute". "Nous décomposons délibérément les choses de milliers de manières différentes pour former une image complète de la façon dont quelque chose fonctionne", explique le professeur de recherche ICREA Ben Lehner, coordinateur du programme de biologie des systèmes au CRG et auteur de l'étude. « C'est comme si vous soupçonniez qu'une bougie est défectueuse, mais au lieu de simplement vérifier cela, le mécanicien démontera toute la voiture et vérifiera toutes les pièces une par une. En analysant dix mille choses à la fois, nous identifions toutes les pièces qui sont vraiment importantes.
Ensuite, nous utilisons des algorithmes d'intelligence artificielle pour interpréter les résultats du laboratoire.
L'un des grands avantages de la méthode, en plus de simplifier le processus nécessaire pour trouver des sites allostériques, est qu'il s'agit d'une technique abordable et accessible pour tout laboratoire de recherche dans le monde. « Cela nécessite simplement l'accès à des réactifs de biologie moléculaire de base, l'accès à un séquenceur d'ADN et à un ordinateur. Avec ces trois composants, n'importe quel laboratoire en 2-3 mois, avec un petit budget, peut réaliser cette expérience sur la protéine d'intérêt qu'il souhaite », assure Júlia Domingo. L'espoir des chercheurs est que nos scientifiques utiliseront la technique pour cartographier rapidement et de manière exhaustive les sites allostériques des protéines humaines, un par un. « Si nous avons suffisamment de données, peut-être qu'un jour nous pourrons aller plus loin et prédire de la séquence protéique à la fonction. Utilisez ces données pour les guider vers de meilleures thérapies pour prédire si un certain changement dans une protéine va dégénérer en maladie », a conclu le chercheur.
