![]() FOLGEN
FOLGEN
Eine neue innovative Technik, die von einem wissenschaftlichen Team des Centre for Genomic Regulation (CRG) in Barcelona entwickelt wurde, hat die Existenz einer Vielzahl von „Fernsteuerungen“ entdeckt, die die Funktion von Proteinen steuern und die als Ziele für wirksamere Medikamente verwendet werden können und wirksam bei verschiedenen Pathologien wie Demenz, Krebs und infektiösen Infektionen.
Diese „Fernsteuerungen“ sind wissenschaftlich als allosterische Stellen bekannt. Das sind Fernbedienungen, die vom Wirkort des Proteins entfernt sind, aber die Fähigkeit haben, es zu regulieren oder zu modulieren“, Júlia Domingo, erste Co-Autorin der Studie, die diesen Mittwoch im Fachblatt „Nature“ erscheint, ABC erklärt. Und er fügt ein Gleichnis hinzu: "Es ist, als könnte man mit dieser Fernbedienung die Glühbirne ein- und ausschalten oder die Intensität des Lichts regulieren."
In diesem Fall, wo es beabsichtigt, die Aktivität von Proteinen zu blockieren oder zu regulieren, die ihre veränderte Funktion im Gefängnis aufrechterhalten. Bei Krebs zum Beispiel haben die Proteine, die eine Mutation erworben haben, ihre Funktion verändert, sie tun dies abnormal und die Zelle wächst ungewöhnlich. In vielen Fällen gibt es keine Medikamente, die diese abnorme Aktivität modulieren oder blockieren können, oder, falls doch, sind sie nicht spezifisch und werden auch von anderen Proteinen freigesetzt, die normal funktionieren.
Traditionell haben Drogenjäger Behandlungen entwickelt, die auf das aktive Zentrum eines Proteins abzielen, dessen kleine Region chemische Reaktionen auslöst, an denen Ziele binden. Der Nachteil dieser Medikamente, die als orthosterische Medikamente bekannt sind, besteht darin, dass die aktiven Stellen vieler Proteine sehr ähnlich sind und die Medikamente viele verschiedene Proteine gleichzeitig gebunden und gehemmt haben, sogar solche, die normal funktionieren und nicht interessant zu berühren sind kann Nebenwirkungen verursachen.
„Dort ging er auf das Konzept der Allosterie und das Potenzial ein, das es hat, um Medikamente zu entwickeln. Das Interessante an allosterischen Stellen ist, dass sie für jedes Protein superspezifisch sind. Wenn diese allosterischen Stellen einen Teil der Proteinoberfläche finden, wo das Medikament landen kann, wird es für dieses Protein extrem spezifisch sein. Wir werden nach wirksameren Medikamenten streben können“, betont der Forscher.
„Wir stellen nicht nur fest, dass diese therapeutischen Stellen reichlich vorhanden sind, sondern es gibt auch Hinweise darauf, dass sie auf viele verschiedene Arten manipuliert werden können. Anstatt sie nur ein- und auszuschalten, können wir ihre Aktivität wie ein Thermostat modulieren. Aus technischer Sicht ist es, als hätten wir Gold gefunden, denn es gibt uns viel Spielraum, um ‚intelligente Medikamente‘ zu entwickeln, die das Schlechte angehen und das Gute überspringen“, erklärt André Faure, Postdoktorand am CRG und erster Co-Autor des Artikels.
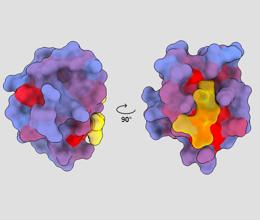 dreidimensionales Bild, das das menschliche Protein PSD95-PDZ3 aus verschiedenen Blickwinkeln zeigt. Ein Molekül, das an das aktive Zentrum bindet, ist gelb dargestellt. Blauer bis roter Farbverlauf weist auf mögliche allosterische Stellen hin – André Faure/ChimeraX
dreidimensionales Bild, das das menschliche Protein PSD95-PDZ3 aus verschiedenen Blickwinkeln zeigt. Ein Molekül, das an das aktive Zentrum bindet, ist gelb dargestellt. Blauer bis roter Farbverlauf weist auf mögliche allosterische Stellen hin – André Faure/ChimeraX
Für diese Entdeckung hat das Team eine Methode verwendet, die es ihnen ermöglicht, eine Protein- und eine systemische Form und eine globale Begegnung mit allen Standorten anzunehmen. Dazu haben sie zwei sehr häufig vorkommende Proteine in unserem menschlichen Proteom ausgewählt. „50 % der Proteinoberfläche haben allosterisches Potenzial. Unsere Methode ermöglicht es, einen Atlas allosterischer Stellen zu erstellen, was die Suche nach wirksamen Medikamenten viel effizienter machen würde“, versichert Júlia Domingo.
Die Autoren der Studie entwickelten eine Technik namens PCA mit doppelter Tiefe (ddPCA), die sie als „Brute-Force-Experiment“ bezeichnen. „Wir brechen Dinge absichtlich auf tausend verschiedene Arten, um ein vollständiges Bild davon zu erhalten, wie etwas funktioniert“, erklärt ICREA-Forschungsprofessor Ben Lehner, Koordinator des Programms „Systems Biology“ am CRG und Autor der Studie. „Es ist, als ob Sie vermuten, dass eine Zündkerze defekt ist, aber anstatt das nur zu überprüfen, nimmt der Mechaniker das ganze Auto auseinander und überprüft alle Teile einzeln. Indem wir zehntausend Dinge auf einmal analysieren, identifizieren wir alle Teile, die wirklich wichtig sind.“
Als nächstes verwenden wir Algorithmen der künstlichen Intelligenz, um die Laborergebnisse zu interpretieren.
Einer der großen Vorteile der Methode besteht neben der Vereinfachung des Prozesses, der zum Auffinden allosterischer Stellen erforderlich ist, darin, dass es sich um eine erschwingliche und zugängliche Technik für jedes Forschungslabor auf der Welt handelt. „Es erfordert lediglich Zugang zu grundlegenden molekularbiologischen Reagenzien, Zugang zu einem DNA-Sequenzierer und einem Computer. Mit diesen drei Komponenten kann jedes Labor in 2-3 Monaten mit einem kleinen Budget dieses Experiment mit dem gewünschten Protein durchführen“, versichert Júlia Domingo. Die Forscher hoffen, dass unsere Wissenschaftler die Technik nutzen, um die allosterischen Stellen menschlicher Proteine schnell und umfassend nacheinander zu kartieren. „Wenn wir genug Daten haben, können wir vielleicht eines Tages einen Schritt weiter gehen und von der Proteinsequenz bis zur Funktion vorhersagen. Verwenden Sie diese Daten, um sie als bessere Therapien zu leiten, um vorherzusagen, ob eine bestimmte Veränderung in einem Protein zu einer Krankheit degenerieren wird“, schloss der Forscher.
